碧云天助我科研路之——半克隆技术首次实现蛋白质功能位点个体水平遗传筛选
编者按:碧云天从成立起就希望“服务科学,造福生命”。我们认为这一理念,需要密切关注科学前沿,更需要关注科学人。为此碧云天推出了“碧云天助我科研路”系列文章,希望能在这个栏目里为大家展示科学家真实的科研和心路历程,有经验、有得失。同时也希望能够提供一个内心交流平台,鼓励正在默默努力的各位。
今天作为第一期,我们很荣幸地邀请到来自中国科学院生物化学与细胞生物学研究所的博士生李庆先生,他将就近日发表在Nature Cell Biology上的文章,与我们分享一下他的科研路。
大家好,我是李庆
现在在中国科学院生物化学与细胞生物学研究所,李劲松研究组攻读博士学位。 今年10月1日,国际知名学术期刊Nature Cell Biology 在线发表了中国科学院生物化学与细胞生物学研究所李劲松和陈勇研究组的最新研究成果“CRISPR–Cas9-mediated base-editing screening in mice identifies DND1 amino acids that are critical for primordial germ cell development”。 作为文章的一作,在此我与各位同行、前辈以及师弟师妹们分享一下我们的研究成果与心得。

我们的工作
利用了最新的CRISPR/Cas9介导的单碱基编辑系统 (BE3) 结合小鼠孤雄单倍体胚胎干细胞介导的半克隆技术,对影响小鼠原始生殖细胞(PGCs)发育的重要基因Dnd1实现个体水平氨基酸功能位点的遗传筛选。CRISPR/Cas9基因编辑系统利用Cas9核酸内切酶结合sgRNA对靶DNA进行切割,诱发DNA进行非同源末端连接(Non-homologous end joining,NHEJ) 或同源重组(Homology-directed repair,HDR)损伤修复机制,进而实现靶基因序列突变(Doudna and Charpentier, 2014)。该技术长期被用于各种细胞系水平的遗传筛选(Chen et al., 2015; Koike-Yusa et al., 2014)。2016年,David R. Liu课题组发表了高效诱导点突变的胞嘧啶单碱基编辑器(BE3), 通过Cas9蛋白上偶联大鼠胞嘧啶脱氨酶(rAPOBEC1),可以将靶位点C•G转变为T•A (Komor et al., 2016)。之后该系统迅速被国内外科学家应用于编辑水稻,小鼠,斑马鱼等模式动物,以及小鼠和人的细胞系(Hess et al., 2017)。单碱基编辑系统除可以进行单个氨基酸位点的基因编辑以外,其更重要的应用可以通过氨基酸位点的遗传筛选进行蛋白结构和功能的研究,进一步可以预测人类疾病中基因重要氨基位点的突变。
我们实验室长期以来一直从事小鼠孤雄单倍体胚胎干细胞(“人造精子”)技术的建立和应用研究。2015年,我们利用CRISPR/
Cas9技术删除“人造精子”中影响胚胎发育的两个重要的印记调控区域,H19-DMR 和 IG-DMR,使得“人造精子”获得了高效产生半克隆小鼠的能力,同时我们通过向“人造精子”中感染全基因组慢病毒文库,证明了“人造精子”技术可以和CRISPR/Cas9技术结合进行个体水平的遗传筛选(Zhong C et al., 2015)。据此,我们推测“人造精子”技术结合BE3系统可以在个体水平进行氨基酸重要功能位点的遗传的筛选。此项工作会加速我们对于蛋白质结构和功能的认识,帮助我们预测疾病的发生。
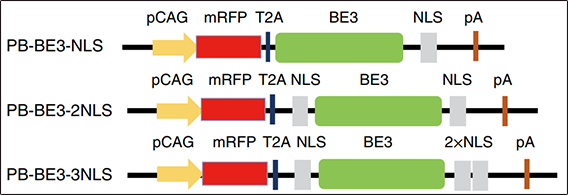
突变效率是进行个体水平遗传筛选最大的障碍,为了改善BE3系统的编辑系统,我们在小鼠胚胎干细胞系(E14)上优化改进了该系统,实现了高效的编辑。我们利用piggyBac转座系统将有强启动子CAG驱动的BE3稳转入ESCs,同时我们发现BE3的N端和C端同时加上一个核定位序列可以进一步提高该系统的编辑效率。随后,我们将该系统应用于小鼠“人造精子”上,发现优化的BE3不仅可以在“人造精子”上实现高效的单碱基编辑,将携带BE3的“人造精子”注入卵子还可以高效地产生纯合点突变的半克隆小鼠。
进一步,
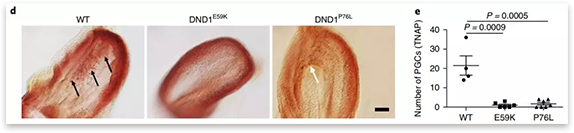
我们设计了一个靶向Dnd1蛋白编码区域的77个sgRNA 的文库,包装成慢病毒文库感染“人造精子”。首先我们在细胞系上证明了这些sgRNA均能很好的编辑对应的突变位点,通过卵胞质内注射这些“人造精子”,在胚胎12.5天检测这些突变对小鼠PGCs发育的影响。通过两轮的遗传筛选,我们发现并证明了DND1蛋白的E59K、V60M、P76L和G82R对PGCs的发育具有重要作用。之后通过和陈勇老师课题组合作,通过蛋白结构的预测我们发现这些突变位点均位于DND1蛋白的RRM1结构域,突变后均可能影响蛋白的稳定性和蛋白-蛋白的相互作用。通过蛋白体外纯化以及细胞系(HEK293T和E14)内过表达实验,均证明了这些突变可能导致蛋白质错误折叠影响了蛋白的稳定性,倾向于被蛋白酶体降解。进一步我们通过CO-IP和GST pull-down 实验证明了这些突变选择性了影响了与NANOS2的结合,从而影响了小鼠PGCs的发育。
我们这个工作以影响小鼠PGCs发育的关键蛋白DND1为例,开创性的证明了单碱基编辑结合“人造精子”技术可以为解析蛋白质关键氨基酸功能提供新方法。该体系的另一潜在的重要应用是筛选的人类疾病相关基因的功能位点,并与已有的SNVs数据库进行比对,预测疾病相关基因的致病位点。随后Nature Cell Biology杂志刊发了费城儿童医院的Ophir Shalem教授的观点文章,高度评价了我们这个工作对于领域内的重要贡献,可以加速个体水平对于疾病分子机制的研究。

2016年5月BE3系统首次出现后,
当时我们本尝试利用该系统直接制备基因编辑的小鼠模型,但是由于购买质粒和前期摸索实验花费了比较多的时间,2017年2月韩国科学家在Nature Biotechnology杂志上首次证明了该系统可以用于小鼠早期胚胎编辑(Kim et al., 2017)。这场比赛中我们已经输了,原本打算完善已有的数据,应该也可以发表一篇够毕业的文章了。但是由于前期我一直在利用我们“人造精子”技术结合CRISPR/Cas9技术筛选PGCs发育过程中新的重要基因,所以我的导师李劲松研究员就不断鼓励我尝试将BE3系统结合“人造精子”技术进行个体水平氨基酸位点的筛选。结合之前积累的相关经验,我们很快就建立了筛选的系统,10月份就获得了阳性的筛选结果,让我们在比赛中扳回一城。
高效的产出离不开个人的勤勉、团队的合作、良好的科研环境,更需要优质的实验试剂与设备。实验过程中,我们使用了碧云天的磷酸钙法细胞转染试剂盒(货号C0508)。依照碧云天详尽的产品使用protocol,我们顺利转染HEK293T细胞并构建了慢病毒sgRNA文库。碧云天一直是我信赖的品牌。品质稳定,价格公道,产品说明十分细致,售后服务专业而贴心。感谢有碧云天相伴,让我在科研路上走得更加稳健。
总之,勤奋和坚持是科学研究必备的技能,专业知识和实验积累是科学研究的利器,良好的科研环境是科学研究的助推器。感谢碧云天给我这次机会,与大家分享我的研究成果与心得,在此也祝各位同行工作顺利。

实验中使用碧云天的产品
References:
Chen, S., Sanjana, Neville E., Zheng, K., Shalem, O., Lee, K., Shi, X., Scott, David A., Song, J., Pan, Jen Q., Weissleder, R., et al. (2015). Genome-wide CRISPR Screen in a Mouse Model of Tumor Growth and Metastasis. Cell 160, 1246-1260.
Doudna, J.A., and Charpentier, E. (2014). The new frontier of genome engineering with CRISPR-Cas9. Science 346, 1-9.
Hess, G.T., Tycko, J., Yao, D., and Bassik, M.C. (2017). Methods and Applications of CRISPR-Mediated Base Editing in Eukaryotic Genomes. Molecular cell 68, 26-43.
Kim, K., Ryu, S.M., Kim, S.T., Baek, G., Kim, D., Lim, K., Chung, E., Kim, S., and Kim, J.S. (2017). Highly efficient RNA-guided base editing in mouse embryos. Nature biotechnology 35, 435-437.
Koike-Yusa, H., Li, Y., Tan, E.P., Velasco-Herrera Mdel, C., and Yusa, K. (2014). Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library. Nature biotechnology 32, 267-273.
Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A., and Liu, D.R. (2016). Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420-424.
Zhong C, Yin Q, Xie Z, Bai M, Dong R, Tang W, Xing Y, Zhang H, Yang S, Chen L, et al. (2015). CRISPR-Cas9-Mediated Genetic Screening in Mice with Haploid Embryonic Stem Cells Carrying a Guide RNA Library. Cell stem cell 17, 1-12.